3.4 Заполнение пропущенных значений в данных
К сожалению, на практике в ходе сбора данных далеко не всегда удается полностью укомплектовать исходные таблицы. Пропуски отдельных значений являются повсеместным явлением и поэтому, прежде чем начать применять статистические методы, обрабатываемые данные следует привести к “каноническому” виду. Для этого необходимо либо удалить фрагменты объектов с недостающими элементами, либо заменить имеющиеся пропуски на некоторые разумные значения.
Проблема “борьбы с пропусками” столь же сложна, как и сама статистика, поскольку в этой области существует впечатляющее множество подходов. В русскоязычных книгах по использованию R (Кабаков, 2014; Мастицкий, Шитиков, 2015) бегло представлены только некоторые функции пакета mice, который, несмотря на свою “продвинутость”, мало удобен для практической работы с данными умеренного и большого объема. Хорошей альтернативой являются методы "knnImpute", "bagImpute" и "medianImpute" функции preProcess() из пакета caret, которую мы рассмотрели в разделе 3.3 как инструмент для трансформации данных.
Используем в качестве примера для дальнейших упражнений таблицу algae, включенную в пакет DMwR и содержащую данные гидробиологических исследований обилия водорослей в различных реках. Каждое из 200 наблюдений содержит информацию о 18 переменных, в том числе:
- три номинальных переменных, описывающих размеры
size = c("large", "medium", "small")и скорость течения рекиspeed = c("high", "low", "medium"), а также время годаseason = c("autumn", "spring", "summer", "winter"), сопряженное с моментом взятия проб; - 8 переменных, составляющих комплекс наблюдаемых гидрохимических показателей: максимальное значение рН
mxPH(1), минимальное содержание кислородаmnO2(2), хлоридыCl(10), нитратыNO3(2), ионы аммонияNH4(2), орто-фосфатыoPO4(2), общий минеральный фосфорPO4(2) и количество хлорофилла аChla(12) (в скобках приведено число пропущенных значений); - средняя численность каждой из 7 групп водорослей
a1-a7(видовой состав не идентифицировался).
Читатель может самостоятельно воспользоваться функциями описательной статистики summary() или describe() из пакета Hmisc, а мы постараемся поддержать добрую традицию и привести парочку примеров диаграмм, построенных с использованием пакета ggplot2 (рис. 3.4):
library(DMwR)
library(ggplot2)
# summary(algae) # вывод не приводится
ggplot(data = algae[!is.na(algae$mnO2), ], aes(speed , mnO2)) +
geom_violin(aes(fill = speed), trim = FALSE,
alpha = 0.3) +
geom_boxplot(aes(fill = speed), width = 0.2,
outlier.colour = NA) +
theme(legend.position = "NA")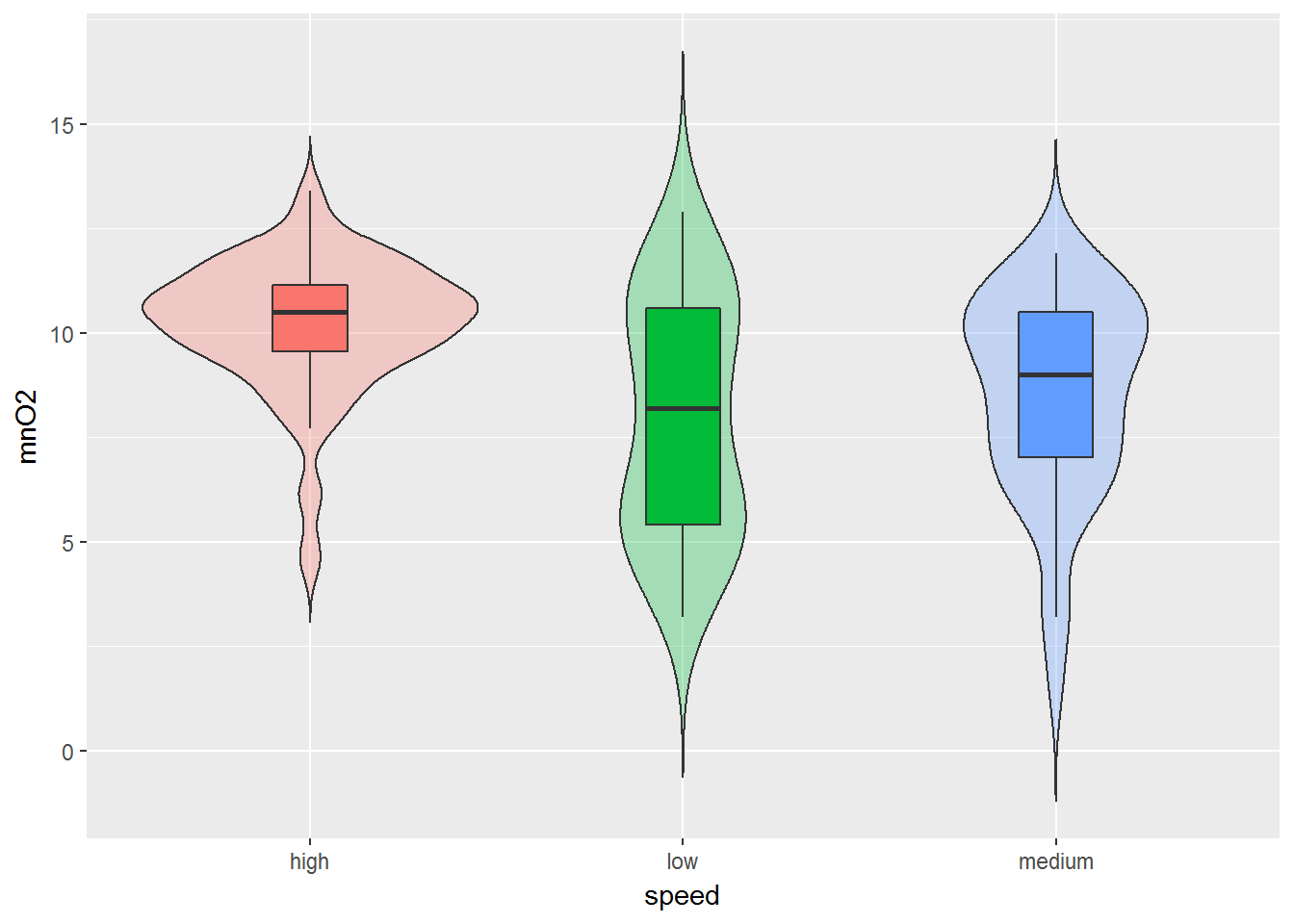
Рисунок 3.4: Распределения значений содержания кислорода в воде рек с разной скоростью течения
На рис. 3.4 мы получили так называемую “скрипичную диаграмму” (violin plot), которая объединяет в себе идеи диаграмм размахов и кривых распределения вероятности. Суть достаточно проста: продольные края “ящиков с усами” (для сравнения приведены тоже) замещаются кривыми плотности вероятности. В итоге, например, легко можно выяснить не только тот факт, что в потоках с быстрым течением (high) содержание кислорода выше, но и ознакомиться с характером распределения соответствующих значений.
Другой пример - категоризованные графики, удобные для визуализации данных, разбитых на отдельные подмножества (категории), каждое из которых отображается в отдельной диаграмме подходящего типа. Такие диаграммы, или “панели” (от англ. panels, facets или multiples), определенным образом упорядочиваются и размещаются на одной странице. Из графиков, представленных на рис. 3.5, легко увидеть, что численность водорослей группы а1 падает с увеличением концентрации фосфатов.
qplot(PO4, a1, data = algae[!is.na(algae$PO4), ]) +
facet_grid(facets = ~ season) +
geom_smooth(color = "red", se = FALSE)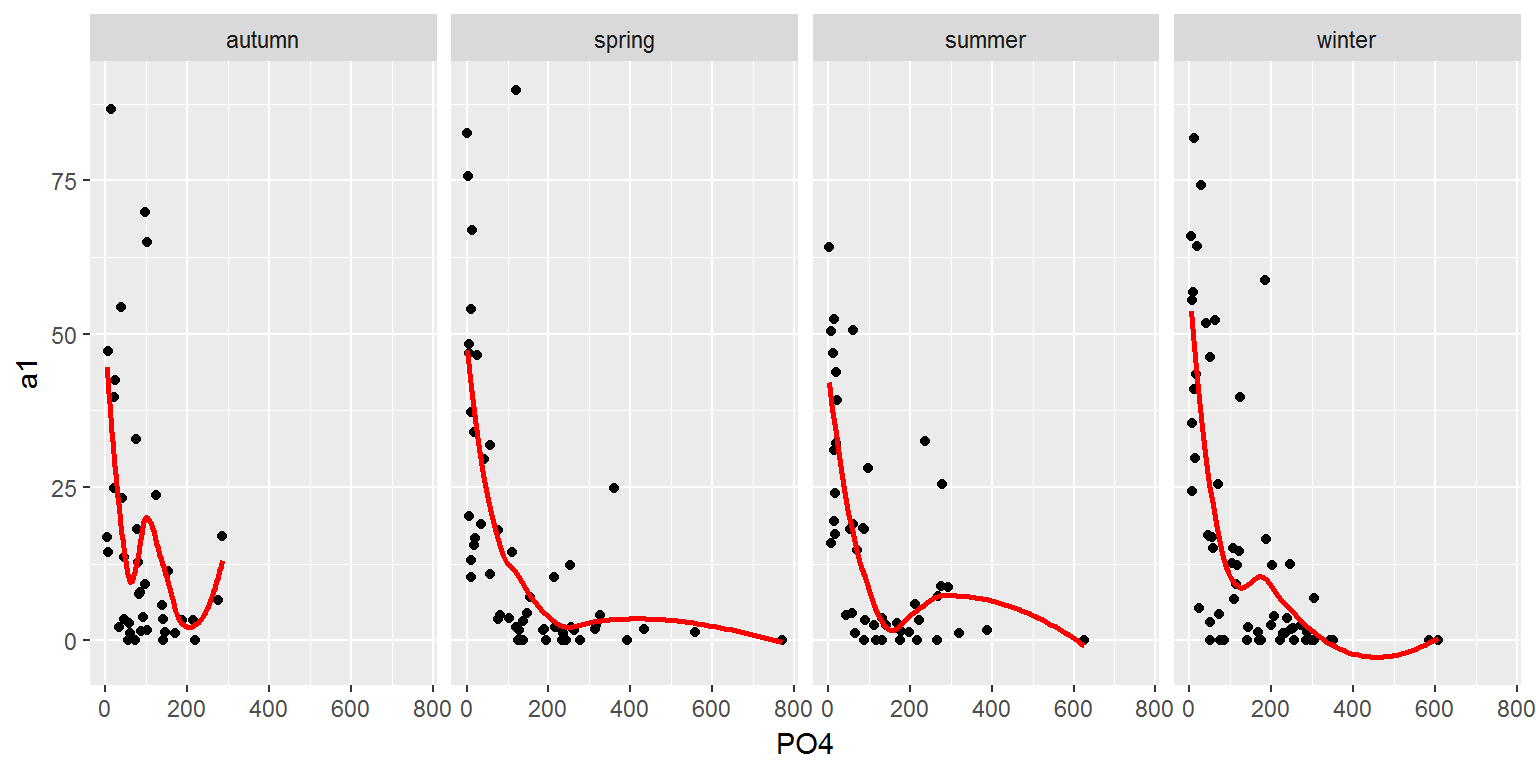
Рисунок 3.5: Графики изменения обилия водорослей в зависимости от содержания минерального фосфора в разное время года
Однако в контексте темы этого раздела важно обратить внимание на то, что мы все время старались блокировать появление пропущенных значений: algae[!is.na(algae$PO4), ]. Если в обрабатываемой таблице обнаружены недостающие данные, то в общих чертах можно избрать одну из следующих возможных стратегий:
- удалить строки с неопределенностями;
- заполнить неизвестные значения выборочными статистиками соответствующей переменной (среднее, медиана и т.д.), полагая, что взаимосвязь между переменными в имеющемся наборе данных отсутствует (это соответствует известному “наивному” подходу);
- заполнить неизвестные значения с учетом корреляции между переменными или меры близости между наблюдениями; постараться обходить эту неприятную ситуацию, используя, например, формальный параметр na.rm некоторых функций.
Последняя альтернатива является наиболее ограничивающей, поскольку она далеко не во всех случаях позволяет осуществить необходимый анализ. В свою очередь, удаление целых строк данных слишком радикально и может привести к серьезным потерям важной информации:
# Число строк с пропущенными значениями:
nrow(algae[!complete.cases(algae),])## [1] 16# Их удаление:
algae <- na.omit(algae)Можно удалить не все строки, а только те, в которых число пропущенных значений превышает, например, 20% от общего числа переменных, для чего существует специальная функция из пакета DMwR:
data(algae)
manyNAs(algae, 0.2)## [1] 62 199algae <- algae[-manyNAs(algae, 0.2), ]В результате мы удалили только две строки (62-ю и 199-ю), где число пропущенных значений больше одного. Обратите внимание, что выполняя команду data(algae), мы каждый раз обновляем в памяти содержимое этого набора данных.
Если мы готовы принять гипотезу о том, что зависимостей между переменными нет, то простым и часто весьма эффективным способом заполнения пропусков является использование средних значений. В том случае, если есть сомнения в нормальности распределения данных, предпочтительнее использовать медиану. Покажем, как это можно сделать с использованием функции preProcess() из пакета caret:
data(algae)
ind <- apply(algae, 1, function(x) sum(is.na(x))) > 0
algae[ind, 4:11]## mxPH mnO2 Cl NO3 NH4 oPO4 PO4 Chla
## 28 6.80 11.1 9.000 0.630 20 4.000 NA 2.70
## 38 8.00 NA 1.450 0.810 10 2.500 3.000 0.30
## 48 NA 12.6 9.000 0.230 10 5.000 6.000 1.10
## 55 6.60 10.8 NA 3.245 10 1.000 6.500 NA
## 56 5.60 11.8 NA 2.220 5 1.000 1.000 NA
## 57 5.70 10.8 NA 2.550 10 1.000 4.000 NA
## 58 6.60 9.5 NA 1.320 20 1.000 6.000 NA
## 59 6.60 10.8 NA 2.640 10 2.000 11.000 NA
## 60 6.60 11.3 NA 4.170 10 1.000 6.000 NA
## 61 6.50 10.4 NA 5.970 10 2.000 14.000 NA
## 62 6.40 NA NA NA NA NA 14.000 NA
## 63 7.83 11.7 4.083 1.328 18 3.333 6.667 NA
## 116 9.70 10.8 0.222 0.406 10 22.444 10.111 NA
## 161 9.00 5.8 NA 0.900 142 102.000 186.000 68.05
## 184 8.00 10.9 9.055 0.825 40 21.083 56.091 NA
## 199 8.00 7.6 NA NA NA NA NA NAlibrary(caret)
pPmI <- preProcess(algae[, 4:11], method = 'medianImpute')
algae[, 4:11] <- predict(pPmI, algae[, 4:11])
(Imp.Med <- algae[ind, 4:11])## mxPH mnO2 Cl NO3 NH4 oPO4 PO4 Chla
## 28 6.80 11.1 9.000 0.630 20.0000 4.000 103.2855 2.700
## 38 8.00 9.8 1.450 0.810 10.0000 2.500 3.0000 0.300
## 48 8.06 12.6 9.000 0.230 10.0000 5.000 6.0000 1.100
## 55 6.60 10.8 32.730 3.245 10.0000 1.000 6.5000 5.475
## 56 5.60 11.8 32.730 2.220 5.0000 1.000 1.0000 5.475
## 57 5.70 10.8 32.730 2.550 10.0000 1.000 4.0000 5.475
## 58 6.60 9.5 32.730 1.320 20.0000 1.000 6.0000 5.475
## 59 6.60 10.8 32.730 2.640 10.0000 2.000 11.0000 5.475
## 60 6.60 11.3 32.730 4.170 10.0000 1.000 6.0000 5.475
## 61 6.50 10.4 32.730 5.970 10.0000 2.000 14.0000 5.475
## 62 6.40 9.8 32.730 2.675 103.1665 40.150 14.0000 5.475
## 63 7.83 11.7 4.083 1.328 18.0000 3.333 6.6670 5.475
## 116 9.70 10.8 0.222 0.406 10.0000 22.444 10.1110 5.475
## 161 9.00 5.8 32.730 0.900 142.0000 102.000 186.0000 68.050
## 184 8.00 10.9 9.055 0.825 40.0000 21.083 56.0910 5.475
## 199 8.00 7.6 32.730 2.675 103.1665 40.150 103.2855 5.475Альтернативой “наивному” подходу является учет структуры связей между переменными. Например, можно воспользоваться тем, что между двумя формами фосфора oPO4 и PO4 существует тесная корреляционная связь. Тогда, например, можно избавиться от некоторых неопределенностей в показателе PO4, вычислив его пропущенные значения по уравнению простой регрессии:
data(algae)
lm(PO4 ~ oPO4, data = algae)##
## Call:
## lm(formula = PO4 ~ oPO4, data = algae)
##
## Coefficients:
## (Intercept) oPO4
## 42.897 1.293# Функция вывода значений PO4 в зависимости от оPO4
fillPO4 <- function(oP) {if (is.na(oP)) return(NA)
else return(42.897 + 1.293 * oP)
}
# Восстановление пропущенных значений PO4
algae[is.na(algae$PO4), 'PO4'] <-
sapply(algae[is.na(algae$PO4), 'oPO4'], fillPO4)
algae[ind, 10]## [1] 48.069 3.000 6.000 6.500 1.000 4.000 6.000 11.000
## [9] 6.000 14.000 14.000 6.667 10.111 186.000 56.091 NAОдно из пропущенных значений удалось восстановить.
Разумеется, легко придти к мысли не утруждать себя перебором всех возможных корреляций, а учесть все связи одновременно и целиком. Использование метода "bagImpute" осуществляет для каждой из имеющихся переменных построение множественной бутстреп-агрегированной модели, или бэггинг-модели (bagging), на основе деревьев регрессии, используя все остальные переменные в качестве предикторов. Этот метод мудр и точен, но требует значительных затрат времени на вычисление, особенно при работе с данными большого объема:
library(ipred)
data(algae)
pPbI <- preProcess(algae[, 4:11], method = 'bagImpute')
algae[, 4:11] <- predict(pPbI, algae[, 4:11])
(Imp.Bag <- algae[ind, 4:11])## mxPH mnO2 Cl NO3 NH4 oPO4 PO4
## 28 6.800000 11.10000 9.00000 0.630000 20.0000 4.00000 30.3218
## 38 8.000000 10.63080 1.45000 0.810000 10.0000 2.50000 3.0000
## 48 8.039073 12.60000 9.00000 0.230000 10.0000 5.00000 6.0000
## 55 6.600000 10.80000 14.55668 3.245000 10.0000 1.00000 6.5000
## 56 5.600000 11.80000 10.04778 2.220000 5.0000 1.00000 1.0000
## 57 5.700000 10.80000 10.04778 2.550000 10.0000 1.00000 4.0000
## 58 6.600000 9.50000 10.04778 1.320000 20.0000 1.00000 6.0000
## 59 6.600000 10.80000 12.40412 2.640000 10.0000 2.00000 11.0000
## 60 6.600000 11.30000 14.55668 4.170000 10.0000 1.00000 6.0000
## 61 6.500000 10.40000 21.51934 5.970000 10.0000 2.00000 14.0000
## 62 6.400000 10.69229 10.04778 1.854339 161.1671 15.08828 14.0000
## 63 7.830000 11.70000 4.08300 1.328000 18.0000 3.33300 6.6670
## 116 9.700000 10.80000 0.22200 0.406000 10.0000 22.44400 10.1110
## 161 9.000000 5.80000 82.65631 0.900000 142.0000 102.00000 186.0000
## 184 8.000000 10.90000 9.05500 0.825000 40.0000 21.08300 56.0910
## 199 8.000000 7.60000 47.50948 4.785011 242.2558 64.94436 181.1578
## Chla
## 28 2.700000
## 38 0.300000
## 48 1.100000
## 55 4.890752
## 56 3.645598
## 57 4.346394
## 58 3.894385
## 59 4.346394
## 60 4.189956
## 61 14.476727
## 62 5.948531
## 63 3.193589
## 116 64.582758
## 161 68.050000
## 184 3.478534
## 199 11.283728Наконец, третий метод функции preProcess() для заполнения пропусков - "knnImpute" - основан на простейшем, но чрезвычайно эффективном алгоритме k ближайших соседей (k-nearest neighbours) или kNN. В основе метода kNN лежит гипотеза о том, что тестируемый объект d будет иметь примерно тот же набор признаков, что и обучающие объекты в локальной области его ближайшего окружения (рис. 3.6).
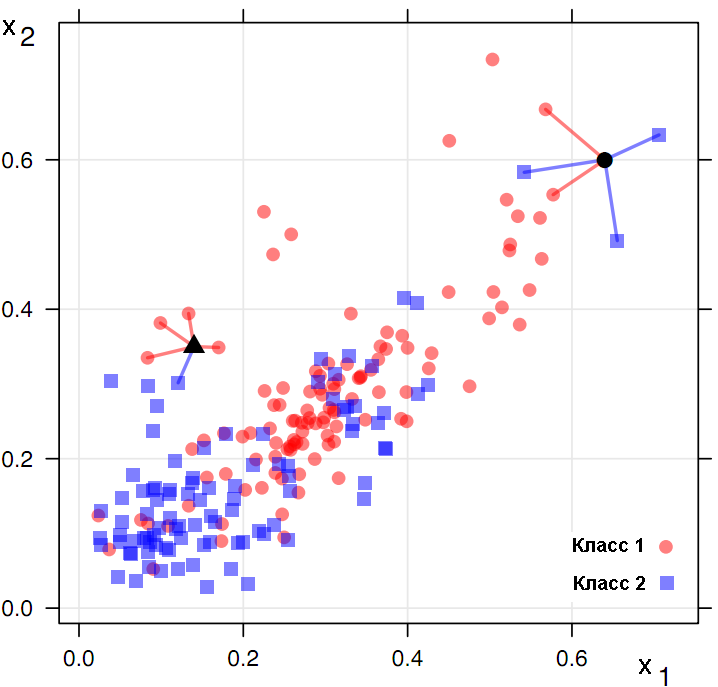
Рисунок 3.6: Интерпретация метода k ближайших соседей
Если речь идет о классификации, то неизвестный класс объекта определяется голосованием \(k\) его ближайших соседей (на рис. 3.6 \(k = 5\)). kNN-регрессия оценивает значение неизвестной координаты \(Y\), усредняя известные ее величины для тех же \(k\) соседних точек.
Одна из важных проблем kNN - выбор метрики, на основе которой оценивается близость объектов. Наиболее общей формулой для подсчета расстояния в m-мерном пространстве между объектами \(\mathbf{X_1}\) и \(\mathbf{X_2}\) является мера Минковского: \[ D_S (\mathbf{X_1}, \mathbf{X_2}) = (\sum |x_{1i} - x_{2i}|^p )^{1/r},\]
где \(i\) изменяется от 1 до \(m\), а \(r\) и \(p\) - задаваемые исследователем параметры, с помощью которых можно осуществить нелинейное масштабирование расстояний между объектами. Мера расстояния по Евклиду получается, если принять в метрике Минковского \(r = p = 2\), и является, по-видимому, наиболее общей мерой расстояния, знакомой всем со школы по теореме Пифагора. При \(r = p = 1\) имеем “манхеттенское расстояние” (или “расстояние городских кварталов”), не столь контрастно оценивающее большие разности координат \(x\). Вторая проблема метода kNN заключается в решении вопроса о том, на мнение какого числа соседей \(k\) нам целесообразно положиться? В свое время мы обсудим этот вопрос детально, а сейчас будем ориентироваться на значение \(k = 5\), используемое по умолчанию:
data(algae)
pPkI <- preProcess(algae[, 4:11], method = 'knnImpute')
alg.stand <- predict(pPkI, algae[, 4:11])Получив в результате применения predict() матрицу переменных с пропущенными значениями, заполненными этим методом, мы с удивлением обнаруживаем, что данные оказались стандартизованными (т.е. центрированными и нормированными на стандартное отклонение). Но а как иначе можно было вычислить меру близости по переменным, измеренным в разных шкалах? Пришлось для возвращения в исходное состояние применить обратную операцию:
m <- pPkI$mean
sd <- pPkI$std
algae[, 4:11] <- t(apply(alg.stand, 1, function (r) m + r * sd))
(Imp.Knn <- algae[ind, 4:11])## mxPH mnO2 Cl NO3 NH4 oPO4 PO4 Chla
## 28 6.800 11.10 9.0000 0.6300 20.000 4.0000 21.2400 2.7000
## 38 8.000 9.92 1.4500 0.8100 10.000 2.5000 3.0000 0.3000
## 48 7.762 12.60 9.0000 0.2300 10.000 5.0000 6.0000 1.1000
## 55 6.600 10.80 7.4974 3.2450 10.000 1.0000 6.5000 3.1800
## 56 5.600 11.80 7.4974 2.2200 5.000 1.0000 1.0000 3.1800
## 57 5.700 10.80 6.0474 2.5500 10.000 1.0000 4.0000 0.8600
## 58 6.600 9.50 5.3422 1.3200 20.000 1.0000 6.0000 0.9000
## 59 6.600 10.80 7.4974 2.6400 10.000 2.0000 11.0000 3.1800
## 60 6.600 11.30 7.7774 4.1700 10.000 1.0000 6.0000 3.9400
## 61 6.500 10.40 11.8864 5.9700 10.000 2.0000 14.0000 4.4000
## 62 6.400 10.24 4.4074 1.1638 60.040 5.0300 14.0000 0.8600
## 63 7.830 11.70 4.0830 1.3280 18.000 3.3330 6.6670 1.0200
## 116 9.700 10.80 0.2220 0.4060 10.000 22.4440 10.1110 5.5000
## 161 9.000 5.80 58.5314 0.9000 142.000 102.0000 186.0000 68.0500
## 184 8.000 10.90 9.0550 0.8250 40.000 21.0830 56.0910 0.9400
## 199 8.000 7.60 59.7922 3.6018 320.908 115.1684 185.9664 12.3958Наконец, зададимся следующим закономерным вопросом: а какой метод лучше? Обычно эта проблема не имеет теоретического решения, и исследователь полагается на собственную интуицию и опыт. Но мы можем оценить, насколько расходятся между собой результаты, полученные каждым способом заполнения. Для этого сформируем блок данных из \(3 \times 16 = 48\) строк исходной таблицы с пропусками, заполненными тремя методами ("Med", "Bag", "Knn"), и выполним снижение размерности пространства переменных методом главных компонент в двумерное (см. раздел 2.4). Посмотрим, как “лягут карты” на плоскости (рис. 3.7):
ImpVal <- rbind(Imp.Med, Imp.Knn)
ImpVal <- rbind(ImpVal, Imp.Bag)
Imp.Metod <- as.factor(c(rep("Med", 16), rep("Knn", 16), rep("Bag", 16)))
library(vegan); library(RANN)
Imp.M <- rda(ImpVal ~ Imp.Metod, ImpVal)
plot(Imp.M, display = "sites", type = "p")
ordihull(Imp.M, Imp.Metod, draw = "polygon", alpha = 67,
lty = 2, col = c(1, 2, 3), label = TRUE)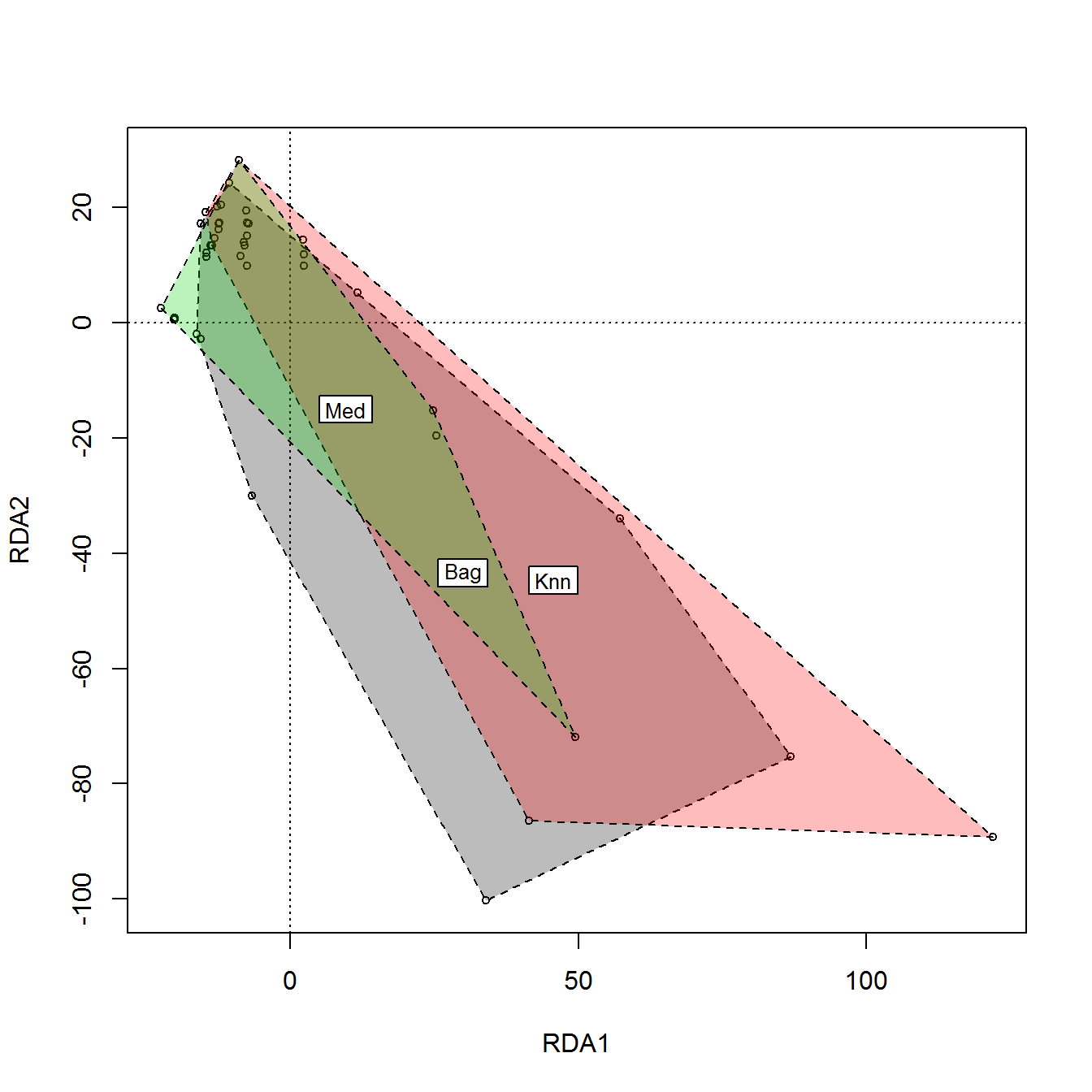
Рисунок 3.7: Ординационная диаграмма блоков данных таблицы algae с пропущенными значениями, заполненными разными способами
На рис. 3.7 мы выделили контуром (hull), проведенным через крайние точки, области каждого из трех блоков данных и поместили метку метода в центры тяжести полученных многоугольников. Понятно, что медианное заполнение характеризуется меньшей вариацией результатов, поскольку игнорирует специфичность свойств каждого объекта. Оба других метода, учитывающих внутреннюю структуру данных, дали приблизительно похожие результаты.