ГЛАВА 10 Кластерный анализ
10.1 Алгоритмы кластеризации, основанные на разделении
Алгоритмы неиерахического разделения (Partitioning algorithms) осуществляют декомпозицию набора данных, состоящего из \(n\) наблюдений, на \(k\) групп (кластеров) с заранее неизвестными параметрами. При этом выполняется поиск центроидов - максимально удаленных друг от друга центров сгущений точек \(C_k\) с минимальным разбросом внутри каждого кластера. К разделяющим алгоритмам относятся:
- метод \(k\) средних Мак-Кина (\(k\)-means clustering; MacQueen, 1967), в котором каждый из \(k\) кластеров представлен центроидом;
- разделение вокруг \(k\) медоидов или PAM (Partitioning Around Medoids; Kaufman, Rousseeuw, 1990), где медоид - это центроид, координаты которого смещены к ближайшему из исходных объектов данных;
- алгоритм CLARA (Clustering Large Applications) - метод, весьма похожий на PAM и используемый для анализа больших наборов данных.
Метод \(k\) средних выполняет кластеризацию следующим образом:
- Назначается число групп (\(k\)), на которые должны быть разбиты данные. Случайно выбирается \(k\) объектов исходного набора как первоначальные центры кластеров.
- Каждому наблюдению присваивается номер группы по самому близкому центроиду, т.е. на основании наименьшего евклидова расстояния между объектом и точкой \(C_k\).
- Пересчитываются координаты центроидов \(\mu_k\) всех \(k\) кластеров и вычисляются внутригрупповые разбросы (within-cluster variation) \(W(C_k) = \sum_{x_i \in C_k} (x_i - \mu_k)^2\). Если набор данных включает \(р\) переменных, то \(\mu_k\) представляет собой вектор средних с \(p\) элементами.
- Минимизируется общий внутригрупповой разброс \(W_{total} = \sum_k W(C_k) \rightarrow \min\), для чего шаги 2 и 3 повторяются многократно, пока назначения групп не прекращают изменяться или не достигнуто заданное число итераций iter.max. Предельное число итераций для минимизации \(W_{total}\), установленное функцией
kmeans()по умолчанию, составляетiter.max = 10.
В разделе 2.6 мы уже подробно разбирали пример кластеризации 50 штатов США по криминогенной обстановке на 5 групп с использованием функции kmeans() из пакета cluster. Пример сопровождается скриптами, графической интерпретацией разбиения и приводятся конкретные вычисленные значения вышеприведенных статистик. В связи с этим в дальнейшем мы будем обсуждать только нюансы практического использования метода.
Объединение в кластеры методом \(k\) средних - очень простой и эффективный алгоритм, имеющий, однако, две существенные проблемы. Во-первых, итоговые результаты чувствительны к начальному случайному выбору центров групп. Возможное решение этой проблемы состоит в многократном выполнении алгоритма с различным случайным назначением начальных центроидов. Итерация с минимальным значением \(W_{total}\) отбирается как конечный вариант кластеризации. Число таких итераций можно задать параметром nstart функции kmeans():
library(cluster)
data("USArrests")
df.stand <- as.data.frame(scale(USArrests))
set.seed(5)
c(kmeans(df.stand, centers = 5, nstart = 1)$tot.withinss,
kmeans(df.stand, centers = 5, nstart = 25)$tot.withinss) ## [1] 51.63029 48.94420Нам удалось за 25 повторов алгоритма несколько уменьшить значение критерия оптимальности. Вторая проблема - необходимость априори задавать фиксированное число кластеров для разбиения, которое, безусловно, далеко не всегда выбирается оптимальным. Поэтому одной из задач кластерного анализа является подбор оптимального значения \(k\), для которой существует несколько версий решения. Метод “локтя” (elbow method) рассматривает характер изменения разброса \(W_{total}\) с увеличением числа групп \(k\). Объединив все \(n\) наблюдений в одну группу, мы имеем наибольшую внутрикластерную дисперсию, которая будет снижаться до 0 при \(k \rightarrow n\). На каком-то этапе можно усмотреть, что снижение этой дисперсии замедляется - на графике это происходит в точке, называемой “локтем” (родственник “каменистой осыпи” для анализа главных компонент). Построить такой график можно в результате прямого перебора, либо с использованием функции fviz_nbclust() из прекрасного пакета factoextra, предназначенного для визуализации результатов кластерного анализа на основе графической системы ggplot2:
k.max <- 15 # максимальное число кластеров
wss <- sapply(1:k.max, function(k){
kmeans(df.stand, k, nstart = 10)$tot.withinss
})
# Вывод не приводится:
# plot(1:k.max, wss, type = "b", pch = 19, frame = FALSE,
# xlab = "Число кластеров K",
# ylab = "Общая внутригрупповая сумма квадратов")
# Формируем график с помощью fviz_nbclust():
library(factoextra)
fviz_nbclust(df.stand, kmeans, method = "wss") +
geom_vline(xintercept = 4, linetype = 2)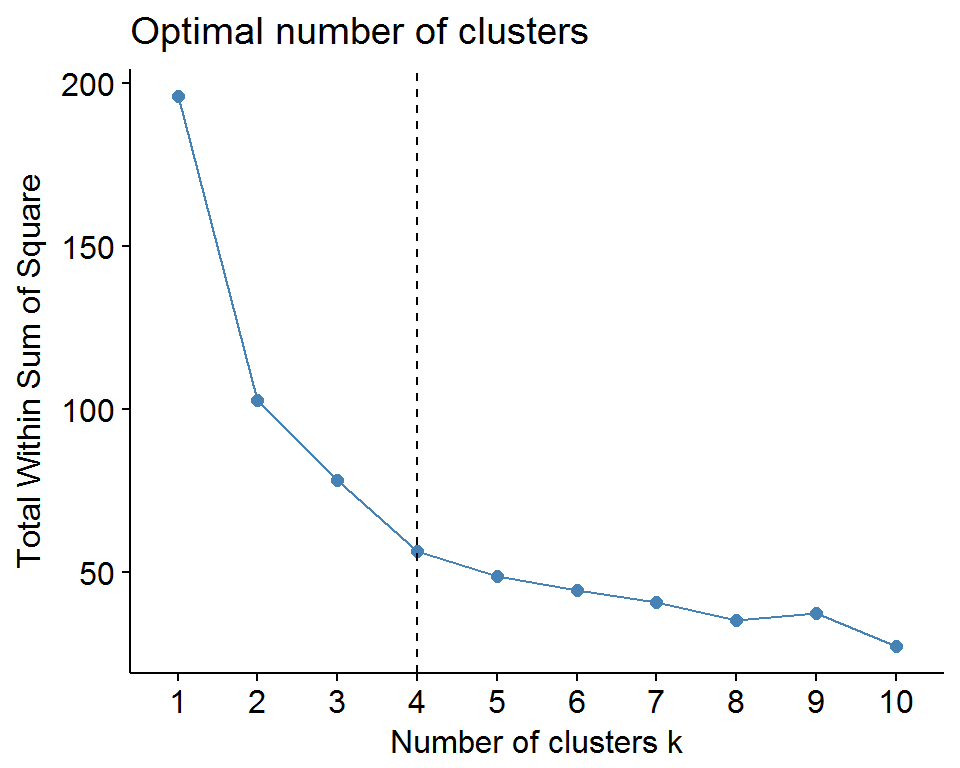
Рисунок 10.1: Выбор оптимального числа кластеров по методу “локтя”
Альтернативой субъективно понимаемым графикам “локтя” является использование статистики разрыва (gap statistic; Tibshirani et al., 2001), которые генерируются на основе ресэмплинга и имитационных процедур Монте-Карло. Пусть \(E_{n}^*\{\log(W_k^*)\}\) обозначает оценку средней дисперсии \(W_k^*\), полученной бутстреп-методом, когда \(k\) кластеров образованы случайными наборами объектов из исходной выборки размером \(n\). Тогда статистика
\[Gap_{n}(k) = E_n^*\{\log(W_k^*)\} - \log(W_k)\]
определяет отклонение наблюдаемой дисперсии \(W_k\) от ее ожидаемой величины при справедливости нулевой гипотезы о том, что исходные данные образуют только один кластер.
При сравнительном анализе последовательности значений \(Gap_n(k), \, k = 2, \dots, K_{max}\) наибольшее значение статистики соответствует наиболее полезной группировке, дисперсия которой максимально меньше внутригрупповой дисперсии кластеров, собранных из случайных объектов исходной выборки:
set.seed(123)
gap_stat <- clusGap(df.stand, FUN = kmeans, nstart = 10, K.max = 10, B = 50)
# Печать и визуализация результатов
print(gap_stat, method = "firstmax")## Clustering Gap statistic ["clusGap"] from call:
## clusGap(x = df.stand, FUNcluster = kmeans, K.max = 10, B = 50, nstart = 10)
## B=50 simulated reference sets, k = 1..10; spaceH0="scaledPCA"
## --> Number of clusters (method 'firstmax'): 4
## logW E.logW gap SE.sim
## [1,] 3.458369 3.633279 0.1749100 0.04956376
## [2,] 3.135112 3.370352 0.2352405 0.04192860
## [3,] 2.977727 3.234819 0.2570927 0.04085179
## [4,] 2.826221 3.120886 0.2946656 0.04153549
## [5,] 2.738868 3.022296 0.2834282 0.04377128
## [6,] 2.669860 2.935234 0.2653739 0.04201917
## [7,] 2.612957 2.855663 0.2427060 0.04029606
## [8,] 2.545027 2.782767 0.2377398 0.04031744
## [9,] 2.471474 2.715964 0.2444898 0.03998257
## [10,] 2.397200 2.654907 0.2577068 0.04074831fviz_gap_stat(gap_stat)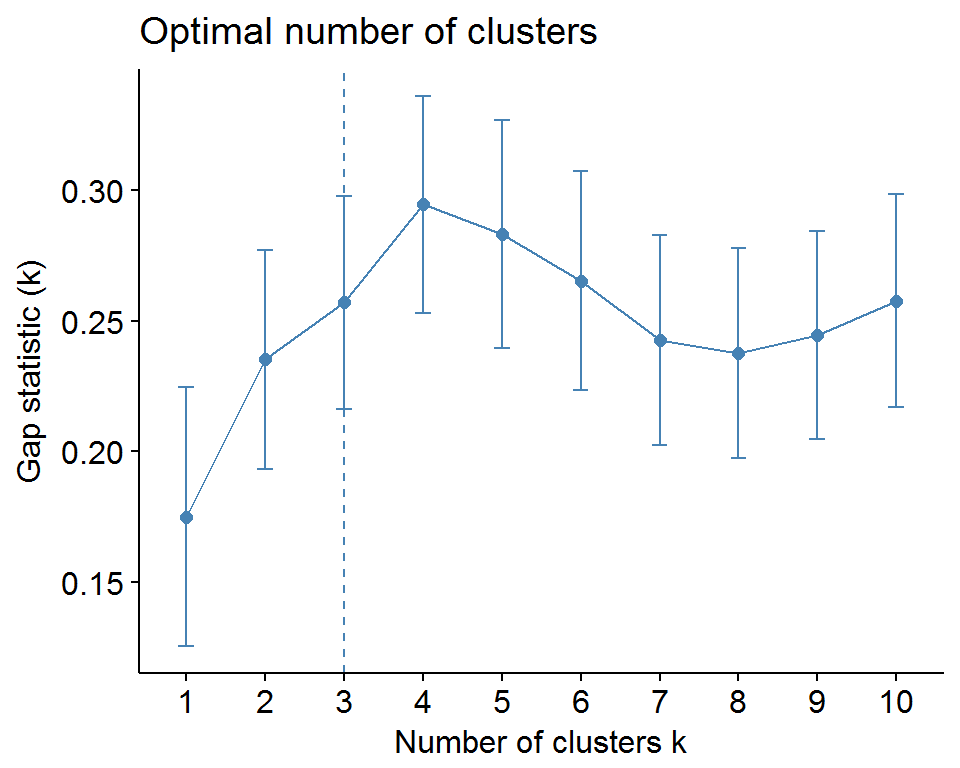
Рисунок 10.2: График GAP-статистики для выбора оптимального числа кластеров
Параметр FUNcluster использованной выше функции clusGap() указывает на необходимый метод кластеризации и при FUN = pam осуществляется разделение вокруг \(k\) медоидов:
set.seed(123)
gap_stat <- clusGap(df.stand, FUN = pam, K.max = 7, B = 100)
print(gap_stat, method = "firstmax")## Clustering Gap statistic ["clusGap"] from call:
## clusGap(x = df.stand, FUNcluster = pam, K.max = 7, B = 100)
## B=100 simulated reference sets, k = 1..7; spaceH0="scaledPCA"
## --> Number of clusters (method 'firstmax'): 4
## logW E.logW gap SE.sim
## [1,] 3.458369 3.632850 0.1744804 0.03737864
## [2,] 3.135112 3.380017 0.2449056 0.04476136
## [3,] 2.981802 3.250681 0.2688793 0.04601583
## [4,] 2.834744 3.141994 0.3072506 0.04709690
## [5,] 2.745099 3.049445 0.3043454 0.04729300
## [6,] 2.685908 2.962081 0.2761727 0.04687566
## [7,] 2.634287 2.886058 0.2517713 0.04560393(k.pam <- pam(df.stand, k = 4))## Medoids:
## ID Murder Assault UrbanPop Rape
## Alabama 1 1.2425641 0.7828393 -0.5209066 -0.003416473
## Michigan 22 0.9900104 1.0108275 0.5844655 1.480613993
## Oklahoma 36 -0.2727580 -0.2371077 0.1699510 -0.131534211
## New Hampshire 29 -1.3059321 -1.3650491 -0.6590781 -1.252564419
## Clustering vector:
## Alabama Alaska Arizona Arkansas California
## 1 2 2 1 2
## Colorado Connecticut Delaware Florida Georgia
## 2 3 3 2 1
## Hawaii Idaho Illinois Indiana Iowa
## 3 4 2 3 4
## Kansas Kentucky Louisiana Maine Maryland
## 3 3 1 4 2
## Massachusetts Michigan Minnesota Mississippi Missouri
## 3 2 4 1 3
## Montana Nebraska Nevada New Hampshire New Jersey
## 3 3 2 4 3
## New Mexico New York North Carolina North Dakota Ohio
## 2 2 1 4 3
## Oklahoma Oregon Pennsylvania Rhode Island South Carolina
## 3 3 3 3 1
## South Dakota Tennessee Texas Utah Vermont
## 4 1 2 3 4
## Virginia Washington West Virginia Wisconsin Wyoming
## 3 3 4 4 3
## Objective function:
## build swap
## 1.035116 1.027102
##
## Available components:
## [1] "medoids" "id.med" "clustering" "objective" "isolation"
## [6] "clusinfo" "silinfo" "diss" "call" "data"Метод PAM во многом идентичен алгоритму \(k\) средних за исключением того, что вместо вычисления центроидов осуществляется поиск \(k\) наиболее представительных объектов (или медоидов) среди анализируемых наблюдений. Внутрикластерный разброс оценивается при этом по манхэттенскому, а не евклидовому расстоянию, как в kmeans(). Мы прибегли к простейшему варианту вычислений - использованию функции pam() из пакета cluster, которая выделила в качестве центральных представителей классов Алабаму, Мичиган, Оклахому и Нью-Гэмпшир.
Поскольку параметром функции pam() также является число кластеров \(k\), проверим оптимальность его значения третьим из наиболее популярных методов - по средней ширине силуэта (average silhouette width). Для каждого найденного кластера может быть вычислена “ширина силуэта”:
\[s_i = \frac{b(i) - a(i)}{\max[b(i), a(i)]},\]
где \(a(i)\) - среднее расстояние между объектами \(i\)-го кластера, \(b(i)\) - среднее расстояние от объектов \(i\)-го кластера до другого кластера, самого близкого к \(i\)-му. Диаграмму силуэтов можно построить с использованием функции fviz_silhouette() из пакета factoextra:
fviz_silhouette(silhouette(k.pam))## cluster size ave.sil.width
## 1 1 8 0.39
## 2 2 12 0.31
## 3 3 20 0.28
## 4 4 10 0.46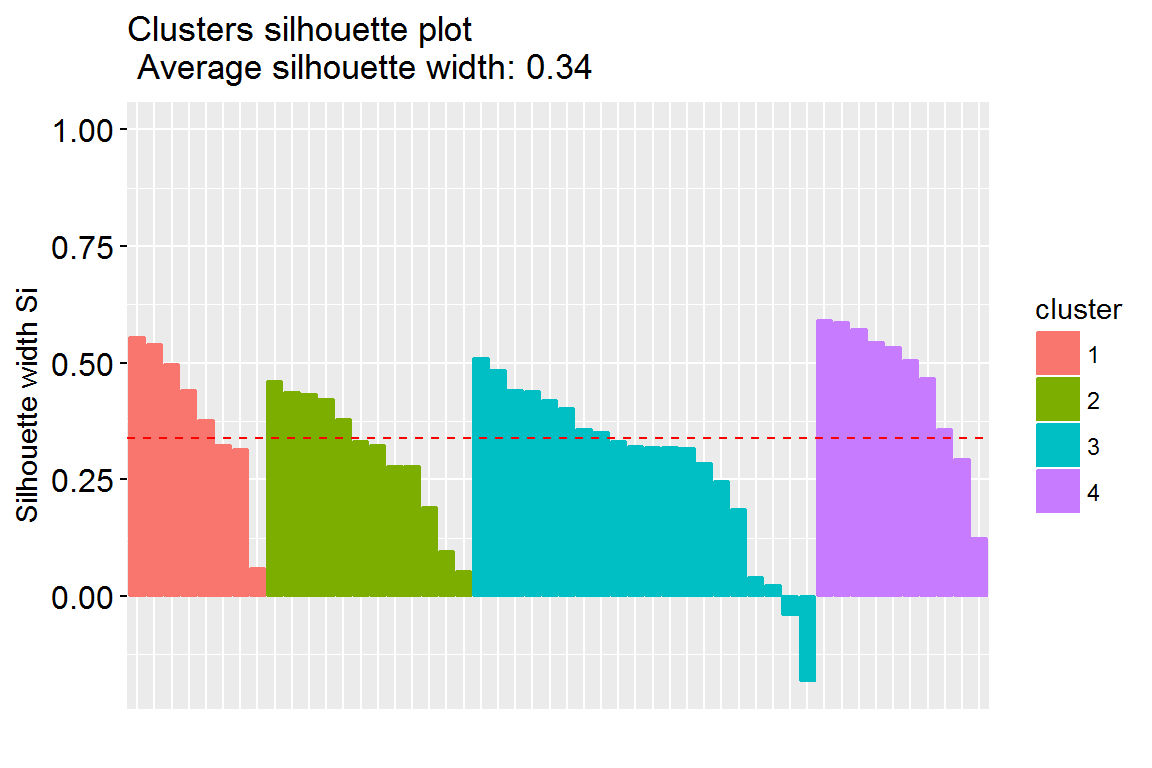
Рисунок 10.3: Диаграмма силуэтов при разбиении на 4 кластера
На рис. 10.3 для каждого из 50 штатов показана разность \(s\) средних расстояний до объектов своего кластера и чужого кластера, ближайшего к анализируемому объекту. Общее среднее из этих значений Smean определяет качество выполненной кластеризации. Функция fviz_nbclust() выполняет сканирование разбиений с различными значениями \(k\), и строит график зависимости \(S_{mean}\) от \(k\), подобный представленным на рис. 10.1-10.2.
fviz_nbclust(df.stand, pam, method = "silhouette")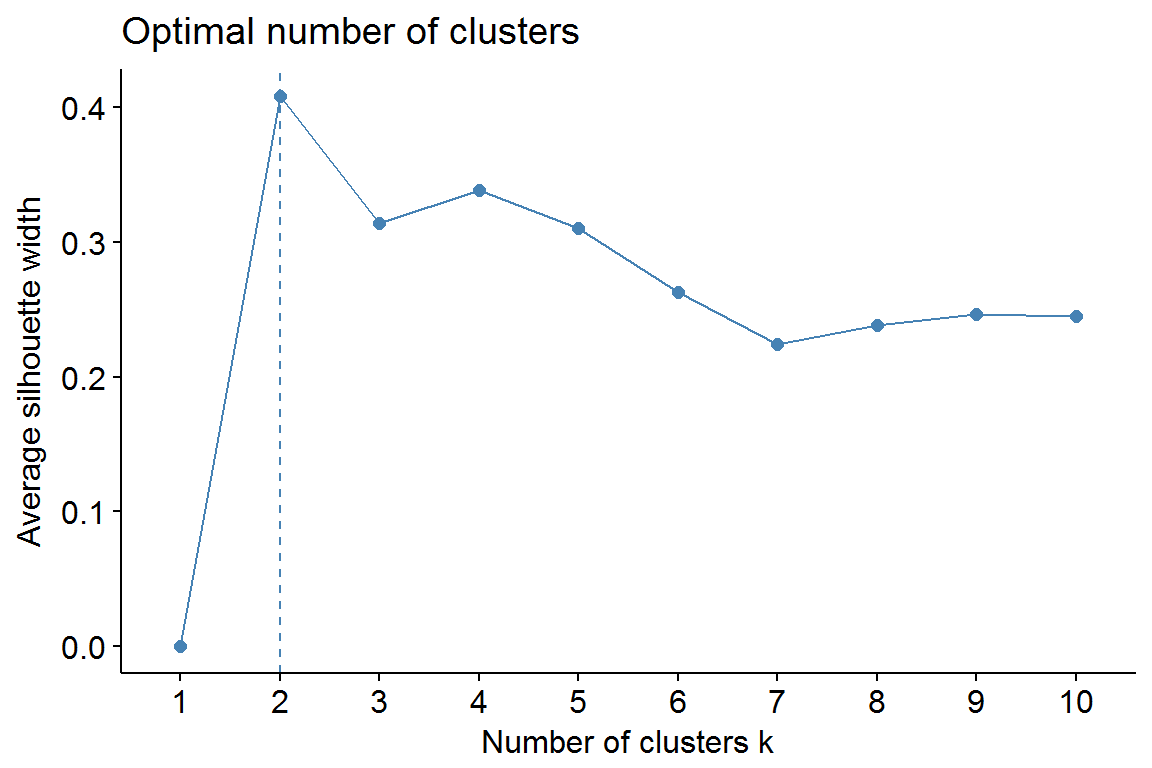
Рисунок 10.4: График ширины силуэтов для выбора оптимального числа кластеров
Метод силуэтов оценил как наилучшее разбиение на два кластера \(k = 2\). Отметим, что в условиях неопределенности различные алгоритмы могут порождать конкурирующие решения. Колеблющийся читатель может в таких случаях пользоваться функцией pamk() из пакета fpc, которая не требует задавать число классов, а оценивает его самостоятельно.
Алгоритм CLARA мало чем отличается от метода PAM, но приспособлен для обработки больших массивов данных (миллионы наблюдений и более). Соответствующая функция clara() из пакета cluster специально оптимизирована для сокращения времени расчетов и рационального использования оперативной памяти.
Значительный интерес представляет построение двумерных диаграмм (ординационных “биплотов”) распределения наблюдений по кластерам, которые формируются с предварительным приведением исходного пространства признаков к двум главным компонентам (см. рис. 2.13 из раздела 2.6). Обычно для этого используется функция clustplot(), но мы предлагаем вниманию читателей функцию fviz_cluster() из пакета factoextra, которая использует для создания диаграмм графическую систему ggplot2.
Эта функция может быть использована для визуализации результатов по методам \(k\) средних, PAM, CLARA и Fanny. Ее простейший формат имеет вид:
fviz_cluster(object, data = NULL, stand = TRUE,
geom = c("point", "text"),
frame = TRUE, frame.type = "convex")где:
object: объект класса"partition", созданный функциямиpam(),clara()илиfanny()из пакетаcluster, или объект, возвращаемый функциейkmeans();data: таблица с исходными данными для кластеризации (этот аргумент необходим только, если используется объектkmeans);stand = TRUE: данные стандартизуются перед выполнением анализа главных компонент;geom: три возможных комбинации стиля отображения наблюдений на графике - текстовые метки"text", точки"point"или оба типа одновременноc("point", "text");frame = TRUE: проводится контур вокруг точек для каждого кластера;frame.type: возможные значения -"convex"или типы эллипсовggplot2::stat_ellipse, включая один из трехc("t", "norm", "euclid").
Построим ординационные диаграммы для результатов кластеризации методами PAM и CLARA. Чтобы получить аккуратные графики, заменим длинные названия штатов США на их сокращенную аббревиатуру из файла St_USA.txt. Во втором случае будем использовать для построения эллипсов многомерное \(t\)-распределение с доверительным интервалом \(\eta = 0.7\):
ShState <- read.delim("data/St_USA.txt")
rownames(df.stand) <- ShState$Short
fviz_cluster(pam(df.stand, 4), stand = FALSE)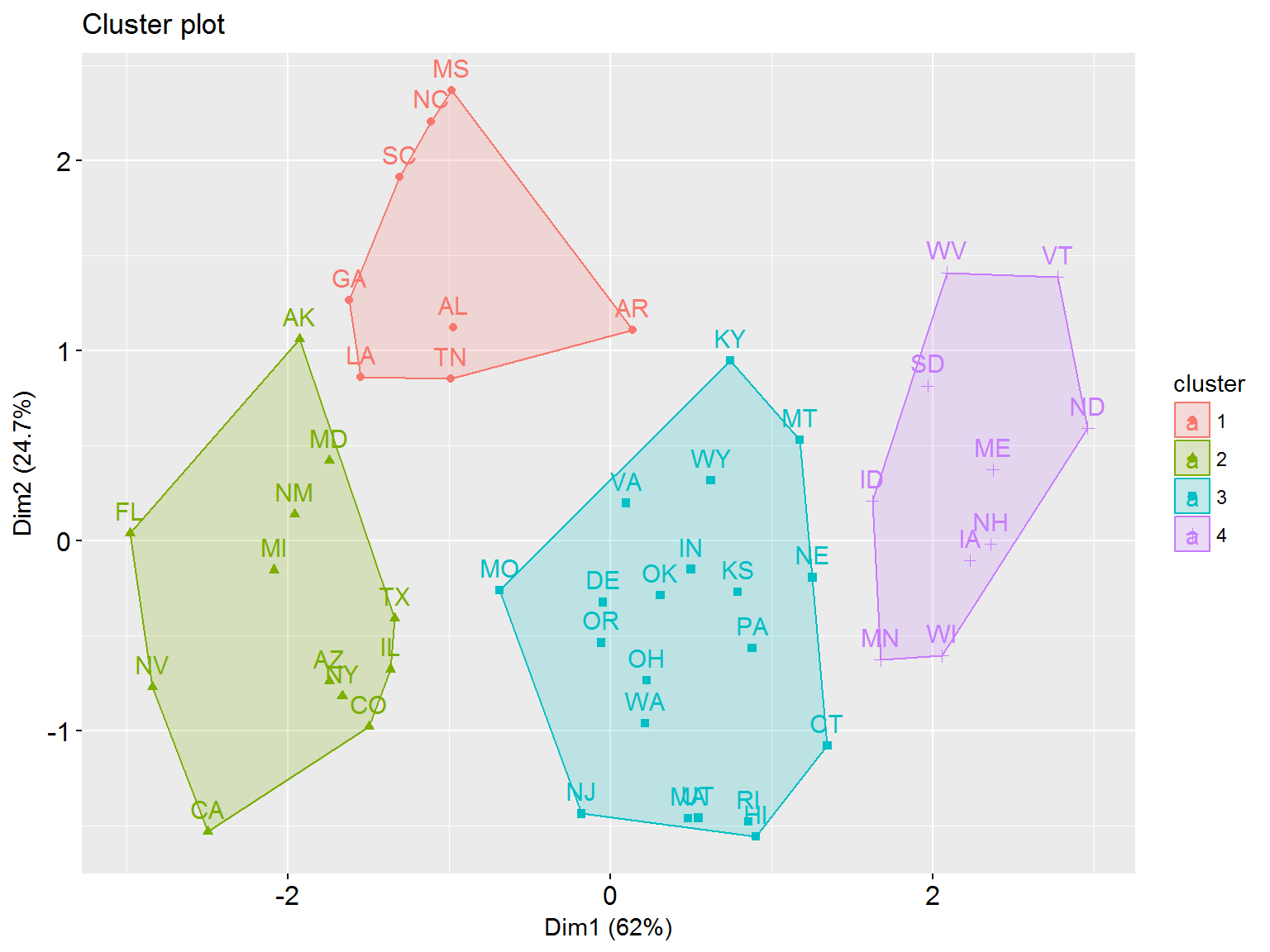
Рисунок 10.5: Диаграмма распределения штатов США по кластерам, полученным методом PAM
fviz_cluster(clara(df.stand, 4), stand = FALSE,
ellipse.type = "t", ellipse.level = 0.7)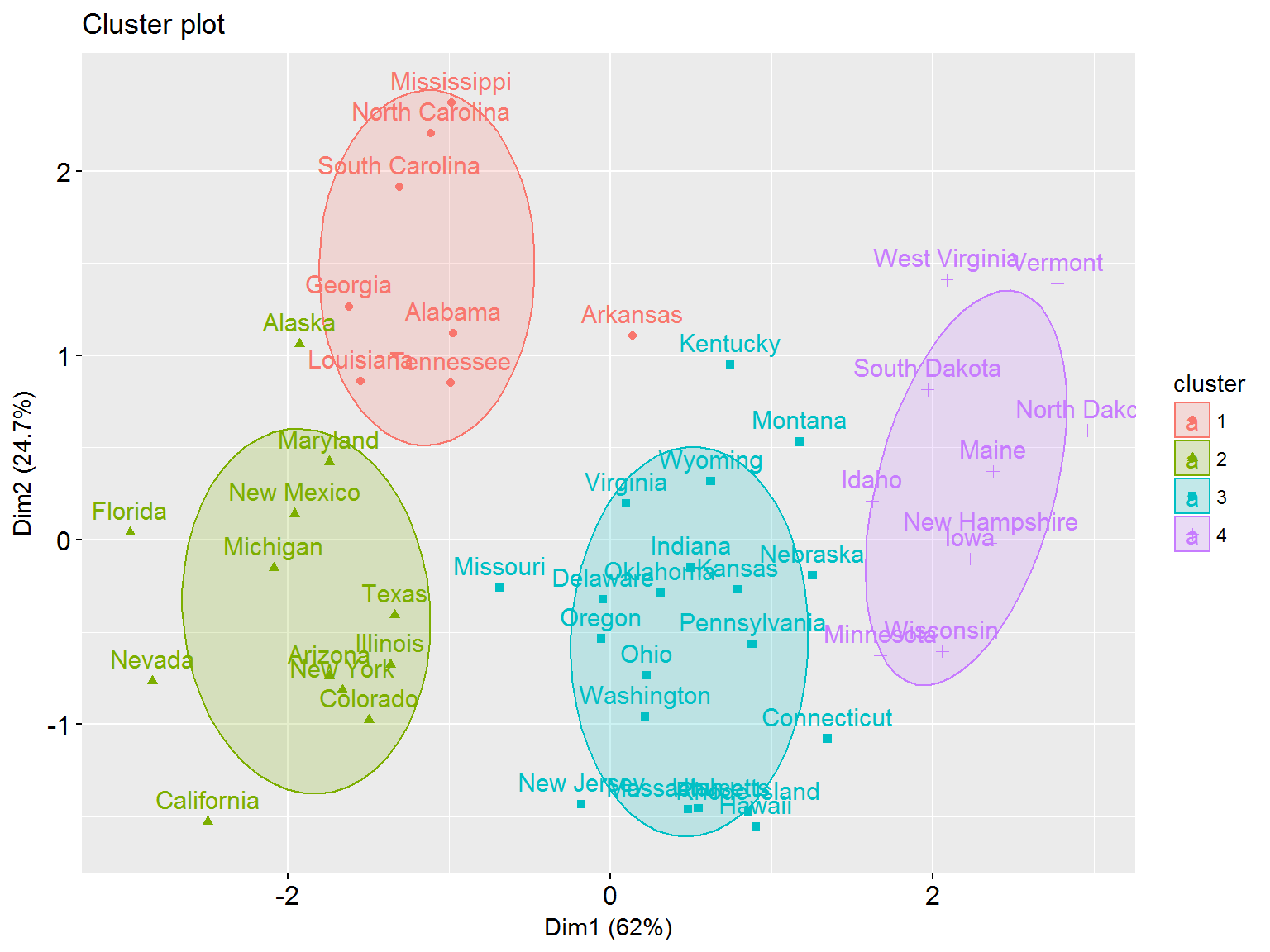
Рисунок 10.6: Диаграмма распределения штатов США по кластерам, полученным методом CLARA; эллипсы построены на основе \(t\)-распределения